今回は、クラスタリングを評価する指標について紹介します。
また、実際にPythonを使用し、クラスタリングを評価する方法を解説します。
レポートやプレゼンでちょうど良いサイズ のグラフを出力するための方法をわかりやすく解説していきます。
| 目的 | 講座名 | 到達点 | レベル/前提 | 想定時間 | こんな人に | 評価 |
|---|---|---|---|---|---|---|
| 現場志向でPython×DSを通しで習得 | 米国データサイエンティストがやさしく教えるデータサイエンスのためのPython講座 | Python基礎〜NumPy/Pandas/ Matplotlib/Seaborn、 Docker+JupyterLabでの環境構築までハンズオン | 初心者〜初級(Mac推奨/軽いプログラミング経験があると◎) | 24時間 | Pythonの基礎固めは完了し、ステップアップをしたいかた | |
| 広く一式を“ブートキャンプ”形式で学ぶ | 【世界で55万人が受講】データサイエンティストを目指すあなたへ〜データサイエンス25時間ブートキャンプ〜 | 統計・Python(Numpy/Pandas)〜機械学習(scikit-learn/TensorFlow)・可視化・Tableauまで網羅 | 初心者OK | 26時間 | データサイエンスとは何をする職業なのか知りたいかた | |
| AI/機械学習の入門(Colab対応・数学やさしめ) | みんなのAI講座 ゼロからPythonで学ぶ人工知能と機械学習 【2025年最新版】 | Python基礎+機械学習の基本(文字認識・株価分析などの小プロジェクト)をColabで体験 | 超初心者〜初級(中学数学レベルで可) | 10時間 | 手を動かしてAIを学習したいかた |
クラスタリングを評価する指標
クラスタリングを評価する指標は大きく2つに分類されます。
- 外部評価指数
正解ラベルありのデータに使用 - 内部評価指数
正解ラベルなしのデータに使用
今回は、正解ラベルありのデータに使用する「外部評価指数」について詳しく紹介していきます。
外部評価指標
外部評価指標には、いくつか指標があります。その中でも主要なものを紹介します。
調整ランド指数(ARI)
調整ランド指数(Adjusted Rand Index, ARI)は、ランド指数(Rand Index, RI)を調整し、偶然による一致を考慮した指標です。
ランド指数とは、データのペアが同じクラスタに属するかを基準に評価する指標のことです。
調整ランド指数の値は、-1から1の範囲であり、1に近いほど性能が良いクラスタリングであることを意味します。
正規化相互情報量(NMI)
正規化相互情報量(Normalized Mutual Information, NMI)は、クラスタリング結果とラベルの情報量を測定します。
正規化相互情報量の値は、0から1の範囲であり、ARI同様に1に近いほど性能が良いクラスタリングであることを意味します。
Python実践 調整ランド指数(ARI)
それでは、Pythonを使用し、ARIを実装する方法を紹介していきます!
scikit-learnライブラリのmetricsをインポートして使用します。
PythonでARIを実装する方法は以下の通りです。
from sklearn.metrics import adjusted_rand_score
ARI = adjusted_rand_score(ラベル, クラスタリング結果)
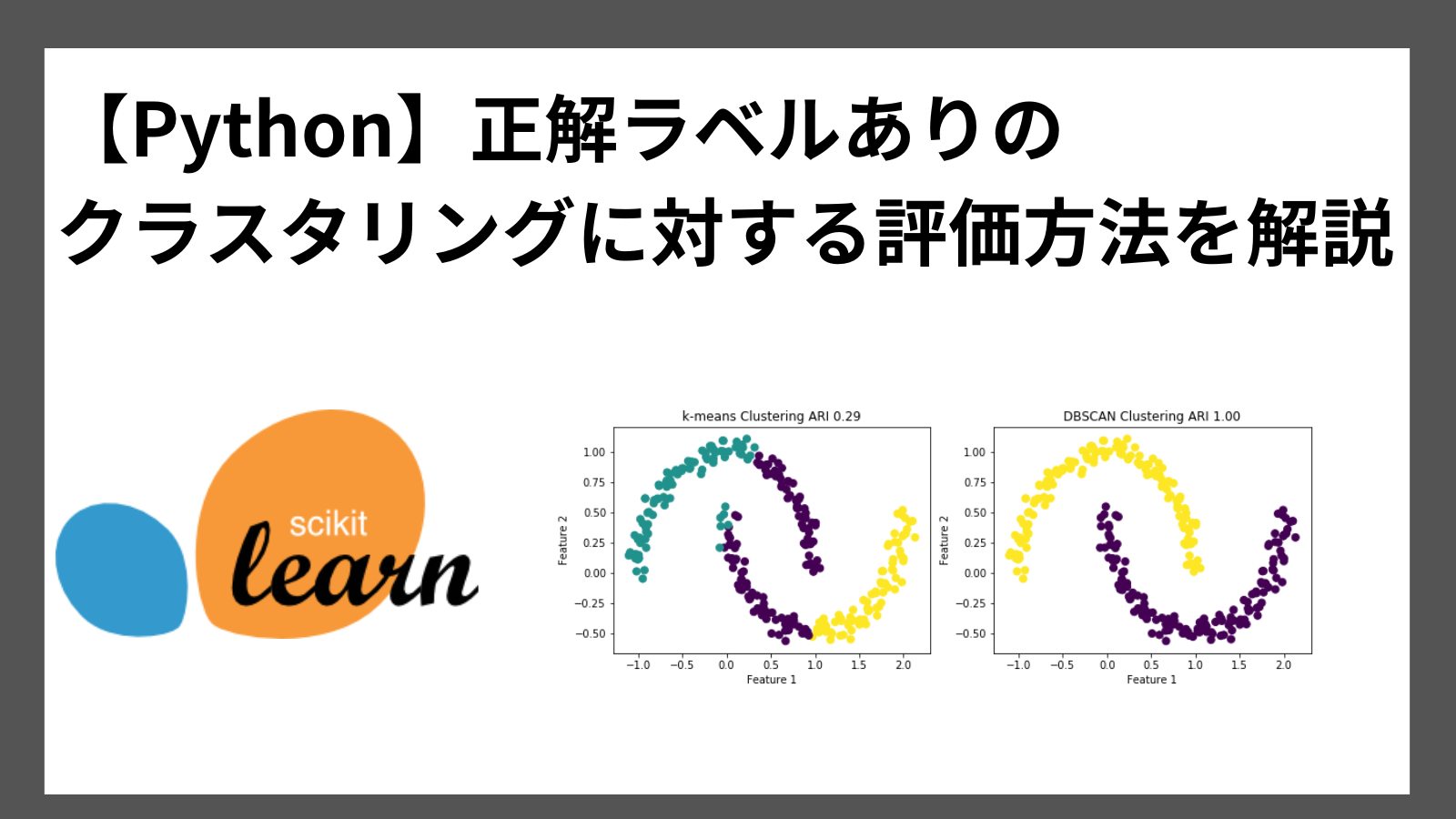
実際にARIを使用し、k-means/DBSCANでクラスタリングした結果を評価してます。
import matplotlib.pyplot as plt
from sklearn.datasets import make_moons
from sklearn.cluster import KMeans, DBSCAN
from sklearn.metrics import adjusted_rand_score
# サンプルデータの生成
X, y = make_moons(n_samples=300, noise=0.05, random_state=42)
# k-meansクラスタリング
kmeans = KMeans(n_clusters=3, random_state=0)
kmeans.fit(X)
labels = kmeans.labels_
# DBSCAN
dbscan = DBSCAN(eps=0.2, min_samples=5)
clusters = dbscan.fit_predict(X)
# 結果の可視化
fig, axes = plt.subplots(1, 2, figsize=(12, 4))
axes[0].scatter(X[:, 0], X[:, 1], c=labels, cmap='viridis', s=50)
axes[0].set_title("k-means Clustering ARI {:.2f}".format(adjusted_rand_score(y, labels)))
axes[0].set_xlabel("Feature 1")
axes[0].set_ylabel("Feature 2")
axes[1].scatter(X[:, 0], X[:, 1], c=clusters, cmap='viridis', s=50)
axes[1].set_title("DBSCAN Clustering ARI {:.2f}".format(adjusted_rand_score(y, clusters)))
axes[1].set_xlabel("Feature 1")
axes[1].set_ylabel("Feature 2")
plt.show()実行結果
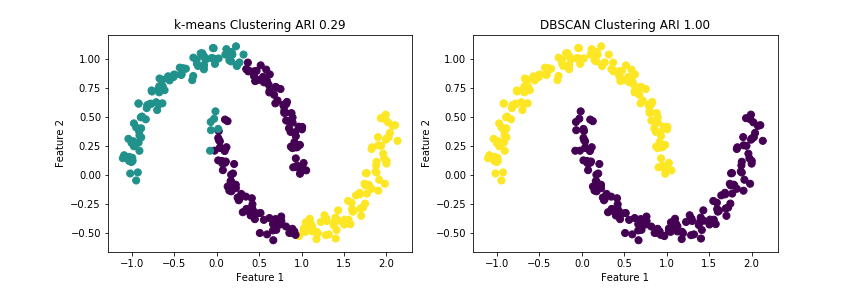
k-meansのARIが0.29なのに対し、DBSCANは1.00であり、今回のデータではDBSCANのほうがクラスタリングの精度が良いことがわかります。
Python実践 正規化相互情報量(NMI)
それでは、Pythonを使用し、NMIを実装する方法を紹介していきます!
scikit-learnライブラリのmetricsをインポートして使用します。
PythonでNMIを実装する方法は以下の通りです。
from sklearn.metrics import normalized_mutual_info_score
NMI = normalized_mutual_info_score(ラベル, クラスタリング結果)
実際にNMIを使用し、k-means/DBSCANでクラスタリングした結果を評価してます。
import matplotlib.pyplot as plt
from sklearn.datasets import make_moons
from sklearn.cluster import KMeans, DBSCAN
from sklearn.metrics import normalized_mutual_info_score
# サンプルデータの生成
X, y = make_moons(n_samples=300, noise=0.05, random_state=42)
# k-meansクラスタリング
kmeans = KMeans(n_clusters=3, random_state=0)
kmeans.fit(X)
labels = kmeans.labels_
# DBSCAN
dbscan = DBSCAN(eps=0.2, min_samples=5)
clusters = dbscan.fit_predict(X)
# 結果の可視化
fig, axes = plt.subplots(1, 2, figsize=(12, 4))
axes[0].scatter(X[:, 0], X[:, 1], c=labels, cmap='viridis', s=50)
axes[0].set_title("k-means Clustering NMI {:.2f}".format(normalized_mutual_info_score(y, labels)))
axes[0].set_xlabel("Feature 1")
axes[0].set_ylabel("Feature 2")
axes[1].scatter(X[:, 0], X[:, 1], c=clusters, cmap='viridis', s=50)
axes[1].set_title("DBSCAN Clustering NMI {:.2f}".format(normalized_mutual_info_score(y, clusters)))
axes[1].set_xlabel("Feature 1")
axes[1].set_ylabel("Feature 2")
plt.show()実行結果
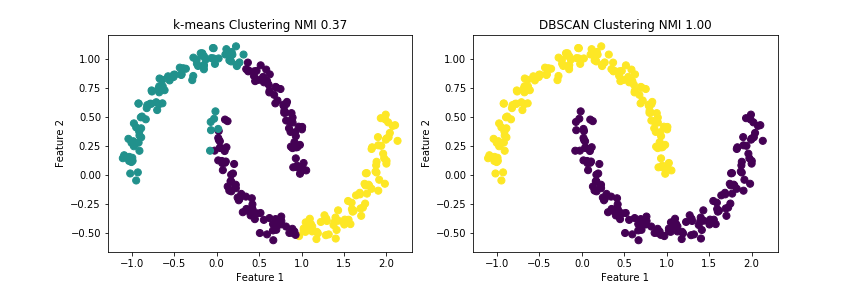
k-meansのNMIが0.37なのに対し、DBSCANは1.00であり、今回のデータではARI同様にDBSCANのほうがクラスタリングの精度が良いことがわかります。
まとめ
クラスタリング結果を評価する方法を紹介しました。
今回は、正解ラベルありのクラスタリングを評価する指標である「外部評価指標」に焦点を当てて説明しました。
また、外部評価指標である「調整ランド指数(ARI)」、「正規化相互情報量(NMI)」について、概要とPythonで実装する方法も合わせて紹介しました。
ここまで読んでくださりありがとうございます。